Abstrait
Contexte/Objectifs : Les vaccins à ARNm synthétiques ont soulevé des inquiétudes concernant l’expression prolongée de la protéine Spike, l’activation immunitaire et les effets hors cible potentiels. Cette étude examine les altérations transcriptomiques chez les personnes présentant de nouveaux événements indésirables ou un cancer après la vaccination par ARNm contre la COVID-19. Méthodes : Un séquençage d’ARN en vrac a été effectué sur le sang périphérique de deux groupes de patients : des personnes présentant de nouveaux événements indésirables non malins et des personnes nouvellement diagnostiquées d’un cancer après la vaccination. Un groupe témoin de personnes en bonne santé a été utilisé à des fins de comparaison. L’expression différentielle des gènes a été analysée à l’aide de DESeq2, et une analyse d’enrichissement des ensembles de gènes (GSEA) a été réalisée à l’aide de la base de données MSigDB et d’ensembles de gènes personnalisés. Résultats : Les deux groupes de patients vaccinés ont présenté une dysrégulation transcriptionnelle généralisée. Dans le groupe des événements indésirables non malins, les enrichissements caractéristiques comprenaient un dysfonctionnement mitochondrial, un stress médié par le protéasome, une instabilité transcriptomique et une inflammation systémique. Le groupe cancéreux a présenté des caractéristiques supplémentaires d’instabilité génomique et de reprogrammation épigénétique. Français La dégradation médiée par le non-sens (NMD), le stress ribosomique et l’activation de MYC étaient prédominants dans les deux groupes, tandis que la signalisation immunitaire via les TLR et les interférons de type I était particulièrement élevée chez les patients atteints de cancer. Conclusions : Les profils transcriptomiques observés indiquent des réponses persistantes au stress cellulaire, un dysfonctionnement mitochondrial et une dysrégulation immunitaire après exposition aux vaccins à ARNm, potentiellement chez les individus sensibles. Des signatures moléculaires partagées et distinctes dans les deux cohortes démontrent des mécanismes sous-jacents contribuant à la symptomatologie et aux complications post-vaccinales, y compris l’oncogenèse et/ou la progression de la maladie maligne. Ces résultats soulignent la nécessité d’une étude plus approfondie de la sécurité à long terme des vaccins à ARNm et de la variabilité de la réponse de l’hôte.

Mots-clés:
vaccin à ARNm ; nouveaux symptômes post-vaccinaux ; transcriptomique ; GSEA ; dysfonctionnement mitochondrial ; protéine de pointe ; dysrégulation immunitaire ; instabilité épigénétique ; événements indésirables ; cancer post-vaccinal
Sujet:
Biologie et sciences de la vie – Biochimie et biologie moléculaire
1. Introduction
Français Les vaccins à ARNm synthétiques ont introduit une nouvelle plateforme dans l’immunisation humaine en codant des antigènes viraux dans des molécules d’ARNm chimiquement modifiées et encapsulées dans des nanoparticules lipidiques. Bien qu’initialement promus pour leur production rapide et leur forte immunogénicité, un nombre croissant de preuves a identifié une série d’événements indésirables, notamment la myocardite, la thrombose, les accidents vasculaires cérébraux, l’amyloïdogenèse, l’arthralgie, les troubles menstruels, les problèmes de reproduction et l’apparition de tumeurs malignes [ 1 , 2 , 3 , 4 , 5 , 6 , 7 ] . Ces événements ont suscité un examen renouvelé des mécanismes moléculaires déclenchés par les constructions d’ARNm synthétiques et de leur devenir intracellulaire [ 8 , 9 ] .
Unlike endogenous mRNA, vaccine-derived transcripts incorporate non-natural features such as N1-methylpseudouridine (m1Ψ) substitution, extended poly(A) tails, and optimized untranslated regions (UTRs) to enhance stability and translational efficiency. These same modifications may alter RNA metabolism and surveillance, contributing to ribosomal infidelity, +1 frameshifting, and defective protein folding [10,11]. Independent studies have reported reverse transcription of vaccine mRNA into DNA via endogenous LINE-1 elements, highlighting issues over genomic integration and persistent expression [10,12]. Recent analyses of BNT162b2 vaccine vials revealed residual plasmid DNA, including SV40 enhancer and antibiotic resistance genes, raising concerns over potential oncogenic risk from unintended genomic exposure [13,14,15,16,17]. Moreover, recent research has shown that spike protein translated from mRNA vaccines can persist in the circulation for extended periods, far beyond initial pharmacokinetic expectations, potentially sustaining inflammatory signaling and immune activation [18,19,20]. Finally, some studies suggest that SARS-CoV-2 bears molecular signatures consistent with synthetic genome assembly, raising further concerns about engineered RNA platforms and their unforeseen biological consequences [21]. These features collectively raise concerns regarding unintended cellular consequences of synthetic mRNA exposure.
Pour combler cette lacune, nous avons réalisé une analyse transcriptomique comparative à partir de données de séquençage d’ARN en vrac issues de sang total de patients présentant des effets indésirables d’apparition récente (groupe 1) et de patients atteints de cancers d’apparition récente (groupe 2) après la vaccination par ARNm contre la COVID-19, ainsi que de témoins sains. Une analyse d’expression différentielle et une analyse d’enrichissement des ensembles de gènes (GSEA) ont été réalisées afin d’identifier les voies moléculaires spécifiques aux marqueurs perturbées dans chaque groupe. En comparant ces deux profils, cette étude vise à mettre en évidence d’éventuelles perturbations transcriptomiques associées à la pathologie liée au vaccin et à explorer si des marqueurs moléculaires distincts ou communs peuvent être à l’origine de différentes trajectoires cliniques post-vaccinales.
2. Materials and Methods
2.1. Caractéristiques de la population de patients et collecte d’échantillons
Nous avons recruté des participants adultes (âgés de 18 à 70 ans) ayant développé de nouveaux symptômes après l’administration de vaccins à ARNm contre la COVID-19 (BNT162b2 [Pfizer] ou ARNm-1273 [Moderna]). Tous les participants ont fourni un consentement éclairé écrit avant leur inclusion dans l’étude. Parmi les personnes symptomatiques présentant des effets indésirables non malins, les manifestations signalées comprenaient des lésions cardiovasculaires, une thrombose, une fatigue chronique et un dysfonctionnement neurologique, apparus peu après la vaccination. Un autre groupe de participants a reçu un diagnostic de cancer comme nouvelle affection peu de temps après l’administration du vaccin à ARNm. La population étudiée a été divisée en deux groupes : le premier était composé de trois personnes ayant développé de nouveaux symptômes post-vaccinaux (groupe 1), et le second comprenait sept personnes ayant reçu un diagnostic de cancer nouvellement diagnostiqué (groupe 2). Une cohorte de 803 personnes en bonne santé a été utilisée comme groupe témoin de référence.
Des échantillons de sang périphérique ont été prélevés conformément aux directives institutionnelles de biosécurité par des infirmières diplômées d’État dans des centres cliniques affiliés à Neo7Bioscience. La ponction veineuse a été réalisée selon une technique stérile standard. Le sang total a été prélevé dans des tubes Streck pré-étiquetés, conservés à 4 °C et transportés le jour même dans des conteneurs réfrigérés au Centre de génomique de l’Université du Nord du Texas pour y être traités.
2.2. Extraction d’ARN
L’ARN total a été extrait à l’aide du kit viral Quick-DNA/RNA (Zymo Research), conformément au protocole d’extraction d’ARN du sang total du fabricant. Toutes les procédures ont été réalisées dans une enceinte de biosécurité de niveau 2 (BSL-2) (Esco Labculture A2). Un tampon de lyse a été ajouté aux échantillons sanguins pour inactiver les agents pathogènes, puis une précipitation à l’éthanol a été effectuée et l’ARN purifié sur colonne. L’ARN élué a été quantifié et conservé à −80 °C jusqu’à la préparation de la banque.
2.3. Préparation de la bibliothèque, synthèse d’ADNc et séquençage à haut débit
Les banques d’ARN ont été préparées à l’aide du kit Illumina TruSeq Stranded Total RNA, intégrant la déplétion de l’ARN ribosomique et la synthèse d’ADNc spécifique de brin. L’ARN a été fragmenté enzymatiquement, rétrotranscrit en ADNc du premier brin et converti en ADNc double brin. Après réparation des extrémités et adénylation, les adaptateurs spécifiques d’Illumina ont été ligaturés et les banques amplifiées par PCR. La qualité et la distribution de la taille des fragments ont été évaluées à l’aide des kits High Sensitivity DNA sur la TapeStation 4200 (Agilent Technologies). Les banques ont été conservées à −20 °C avant le séquençage.
Les bibliothèques normalisées et groupées ont été séquencées sur la plateforme Illumina NextSeq 550 à l’aide du kit NextSeq 500/550 High Output Kit v2.5 (300 cycles), avec un objectif de couverture de 200× par échantillon. PhiX a été ajouté à 1 % comme contrôle interne. Les lectures de séquençage appariées ont été collectées et les indicateurs de qualité du séquençage (par exemple, scores Q30, densité des clusters) ont été surveillés en temps réel grâce au visualiseur d’analyse de séquençage d’Illumina.
2.4. Prétraitement des données, contrôle qualité et alignement
Les données de séquençage brutes, sous forme de fichiers Fastq appariés, ont d’abord été organisées par type d’échantillon et de lecture (R1 et R2). Ces fichiers Fastq ont été stockés dans un répertoire dédié, et la commande cat a été utilisée pour les regrouper en fonction de leurs identifiants d’échantillon. Les fichiers R1 et R2 de chaque échantillon ont ensuite été fusionnés en un seul fichier pour chaque type de lecture, ce qui a permis d’obtenir des fichiers fusionnés en sens inverse et en sens inverse, prêts pour l’analyse en aval.
Des contrôles qualité ont été effectués avant l’étape d’alignement afin de garantir l’intégrité des données. Les lectures de séquençage ont été alignées sur le génome humain de référence (hg38) à l’aide de STAR [ 22 ] , un aligneur largement utilisé pour les données de séquençage d’ARN. STAR gère efficacement l’alignement des lectures dans un environnement multithread, optimisant ainsi le temps de traitement. Les lectures alignées ont ensuite été générées sous forme de fichiers BAM, triées par coordonnées, ce qui est une pratique courante pour les étapes d’analyse ultérieures.
2.5. Analyse d’expression différentielle et visualisation du diagramme volcanique
Français Pour étudier les altérations transcriptomiques associées aux résultats liés au vaccin à ARNm, nous avons effectué deux analyses d’expression différentielle distinctes, chacune comparant un groupe de patients à un groupe commun de témoins sains (n = 803). La première comparaison incluait des individus du groupe 1, qui ont développé des symptômes d’apparition récente non malins après la vaccination par ARNm (n = 3), tandis que la seconde comprenait des individus du groupe 2, diagnostiqués avec un cancer comme une affection d’apparition récente peu après la vaccination (n = 7). Pour les deux analyses, le nombre brut de gènes généré à partir de Salmon [ 23 ] a été entré dans DESeq2 [ 24 ] , qui a appliqué une normalisation interne et modélisé la dispersion en utilisant la distribution binomiale négative. La réduction des changements de repli log₂ a été réalisée en utilisant la méthode « apeglm » pour améliorer l’estimation de la taille de l’effet, en particulier pour les gènes avec un faible nombre de lectures.
Afin de visualiser les schémas globaux d’expression génétique, des graphiques en volcan ont été générés pour chaque comparaison. Dans ces graphiques, l’axe des abscisses représente le log₂ fold change, et l’axe des ordonnées représente le log₁₀ négatif de la valeur p ajustée. Les gènes avec un log₂ fold change supérieur à +1 et une valeur p ajustée (padj) inférieure à 0,05 étaient considérés comme significativement régulés à la hausse et étaient colorés en rouge, tandis que ceux avec un log₂ fold change inférieur à -1 et padj < 0,05 étaient considérés comme significativement régulés à la baisse et étaient colorés en bleu. Tous les gènes restants ne répondant pas à ces critères étaient affichés en gris.
2.6. Analyse d’enrichissement des ensembles de gènes
Pour étudier les perturbations transcriptomiques associées à l’exposition au vaccin à ARNm, nous avons analysé les données d’ARN-seq de trois individus présentant des effets indésirables liés au vaccin et de 803 témoins sains [ 25 ] . Une analyse différentielle de l’expression génique a été réalisée à l’aide de DESeq2 [ 24 ] , et les gènes ont été classés par changement de repli log₂ pour générer le fichier d’entrée pour l’analyse d’enrichissement.
Nous avons ensuite réalisé une analyse d’enrichissement des ensembles de gènes (GSEA) en utilisant le mode pré-classé de l’outil GSEA 4.4.0 du Broad Institute [ 26 ] . Cette approche a été choisie en raison du déséquilibre significatif des classes (3 contre 803 échantillons), car elle améliore la stabilité et l’interprétabilité des résultats d’enrichissement. La statistique d’enrichissement « pondérée » a été appliquée pour intégrer à la fois le classement des gènes et l’ampleur de l’expression, améliorant ainsi la sensibilité aux perturbations biologiquement pertinentes. Nous avons utilisé 1 000 permutations d’ensembles de gènes pour estimer la signification statistique et sélectionné l’option « Pas de réduction » pour conserver les symboles génétiques sans mappage d’alias.
Français La GSEA a été systématiquement menée sur plusieurs collections MSigDB [ 27 ] : H (Hallmark), C2 (KEGG et REACTOME), C5 (GOBP), C7 (Signatures immunologiques) et des ensembles de gènes personnalisés, y compris Gavish et des signatures d’expression dérivées de GSE organisées. Les résultats d’enrichissement ont été interprétés en fonction du score d’enrichissement normalisé (NES) et du taux de fausses découvertes (valeur q du FDR), en sélectionnant les ensembles de gènes qui étaient fortement enrichis (NES positif) ou supprimés (NES négatif), ce qui indiquait respectivement des fonctions biologiques régulées à la hausse ou à la baisse. Pour l’analyse en aval, seuls les ensembles de gènes avec un FDR < 0,25 et un NES absolu ≥ 1,5 ont été retenus. Les ensembles de gènes les plus enrichis et supprimés ont été regroupés manuellement dans des catégories de voies moléculaires d’ordre supérieur.
Afin d’améliorer la pertinence biologique, nous avons exclu les groupes de gènes associés aux tissus non sanguins (par exemple, cerveau, rétine, peau) et au développement embryonnaire, car ils sont peu susceptibles de refléter l’activité transcriptionnelle dans le sang périphérique. Ce filtrage a minimisé le bruit et concentré l’analyse sur les voies liées aux réponses immunitaires, inflammatoires et systémiques.
2.7. Carte des interactions protéine-protéine
Français Pour construire les réseaux de gènes, les informations sur les interactions des gènes différentiellement exprimés à partir de la base de données STRING ont été chargées sur la plateforme logicielle Cytoscape [ 28 , 29 ] . STRING est une base de données d’interactions protéine-protéine connues et prédites qui comprend à la fois les associations protéiques physiques et fonctionnelles. La base de données STRING couvre actuellement 24 584 628 protéines provenant de 5 090 organismes [ 30 ] . STRING génère un réseau à partir d’une liste d’entrée de protéines basée sur des associations provenant de diverses sources de données, notamment des prédictions de contexte génomique, des expériences de laboratoire à haut débit, l’exploration de texte automatisée et les connaissances antérieures dans les bases de données [ 31 ] . Le réseau peut être visualisé dans STRING ou exporté pour visualisation et analyse en dehors de STRING ; par exemple, le réseau peut être exporté directement vers Cytoscape. Cytoscape combine des réseaux d’interactions biomoléculaires avec des données d’expression à haut débit et d’autres états moléculaires dans un modèle de réseau conceptuel intégré.
3. Résultats
Afin de caractériser l’impact transcriptionnel global de la vaccination par ARNm chez les personnes présentant des effets indésirables post-vaccinaux, nous avons réalisé des analyses d’expression génique différentielle comparant chaque groupe de patients à la cohorte témoin saine. Comme le montre la figure 1 , les graphiques en volcan visualisent la distribution des changements d’expression génique pour le groupe 1 (apparition de nouveaux symptômes post-vaccinaux non malins) et le groupe 2 (apparition de nouveaux diagnostics de cancer). Dans les deux groupes, un net décalage transcriptomique est observé, avec des centaines de gènes présentant une dysrégulation significative. Le groupe cancéreux présente notamment une distribution plus large de gènes sous-exprimés, dont plusieurs présentent des variations d’amplitude extrêmes, suggérant une suppression profonde des voies de régulation clés.
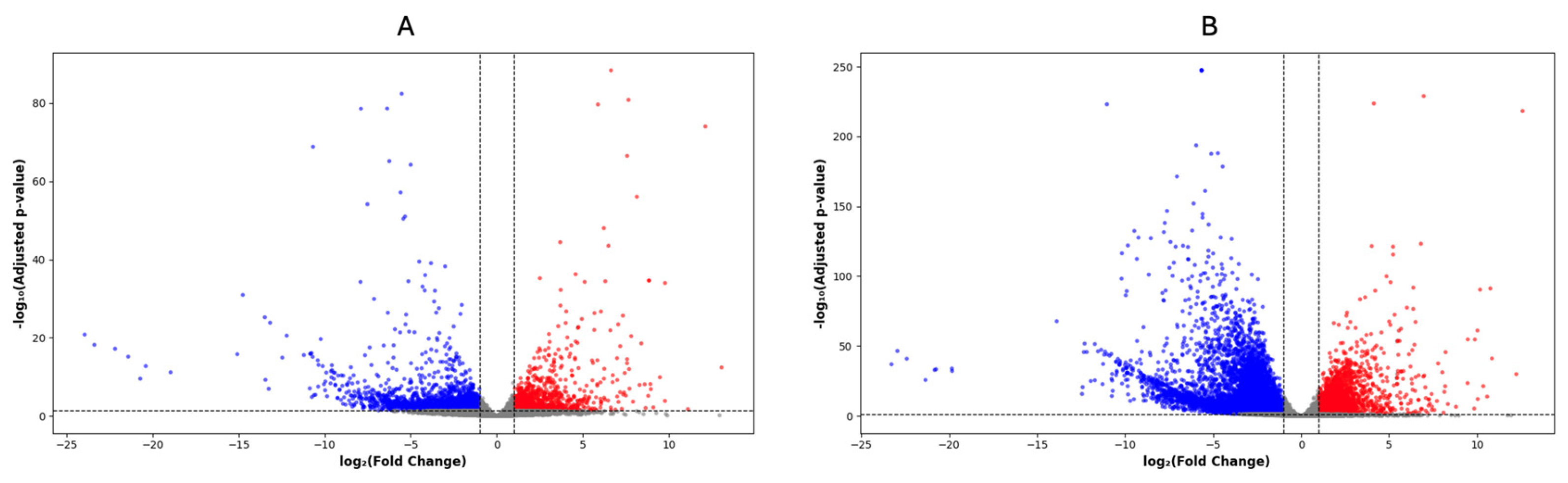
Figure 1. Profils d’expression différentielle à l’échelle du transcriptome chez les personnes vaccinées. ( A ) Graphique en volcan montrant l’expression différentielle des gènes chez les personnes présentant des effets indésirables d’apparition récente après la vaccination par ARNm contre la COVID-19 (n = 3) par rapport aux témoins sains (n = 803). ( B ) Graphique en volcan montrant l’expression différentielle des gènes chez les personnes diagnostiquées avec un cancer d’apparition récente peu après avoir reçu la vaccination par ARNm contre la COVID-19 (n = 7) par rapport à la même cohorte témoin. Chaque point représente un seul gène tracé par le changement de facteur log₂ (axe des x) et la valeur de p ajustée de –log₁₀ (axe des y). Les gènes avec une régulation positive significative (log₂FC > 1, padj < 0,05) sont marqués en rouge, tandis que les gènes significativement régulés à la baisse (log₂FC < –1, padj < 0,05) sont indiqués en bleu. Les gènes non significatifs apparaissent en gris. Ces graphiques révèlent une dysrégulation transcriptionnelle généralisée dans les deux groupes de patients, servant de base à l’analyse ultérieure de l’enrichissement des voies.
Une analyse d’enrichissement des ensembles de gènes (GSEA) a été réalisée séparément pour les deux groupes d’étude : le groupe 1, composé de personnes présentant de nouveaux effets indésirables après la vaccination par ARNm contre la COVID-19, et le groupe 2, composé de personnes diagnostiquées d’un cancer d’apparition récente après la vaccination, afin de caractériser les perturbations transcriptomiques associées à l’exposition au vaccin. L’analyse GSEA a été réalisée à l’aide des collections d’ensembles de gènes prédéfinies de la base de données MSigDB, et les ensembles de gènes enrichis ont été systématiquement regroupés en fonction de leur pertinence pour les voies moléculaires d’ordre supérieur. Ce regroupement basé sur les voies a permis une interprétation intégrative des altérations transcriptomiques dans tous les domaines de la biologie des systèmes.
Français Dans le groupe 1, les ensembles de gènes les plus enrichis se sont regroupés en six catégories clés : (1) Dysfonctionnement du transport d’électrons mitochondrial et espèces réactives de l’oxygène (ROS), (2) Stress de dégradation des protéines médié par le protéasome, (3) Instabilité transcriptomique et stress translationnel, (4) Réponse inflammatoire et immunitaire systémique, (5) Dysfonctionnement de l’endothélium et (6) Signalisation proliférative et suppression du contrôle tumoral. En revanche, le groupe 2 a présenté un modèle d’enrichissement distinct mais chevauchant. Les caractéristiques identifiées dans ce groupe comprennent : (1) Instabilité transcriptomique et stress translationnel, (2) Réponse inflammatoire et immunitaire systémique, (3) Dysfonctionnement de l’endothélium, (4) Signalisation proliférative et suppression du contrôle tumoral et (5) Instabilité génomique et décalage épigénétique. Comme le montre le tableau 1 , plusieurs ensembles de gènes au sein de chaque catégorie de marqueurs ont montré des scores d’enrichissement élevés (NES > 1,5 ou < –1,5) et des valeurs q FDR statistiquement significatives (FDR < 0,05), étayant la présence d’une dysrégulation moléculaire coordonnée. Des courbes représentatives du score d’enrichissement (ES) sont fournies dans le supplément. La figure S1 illustre la distribution des rangs de gènes contribuant à chaque caractéristique.
Tableau 1. Résultats de l’enrichissement des ensembles de gènes classés par perturbations des voies moléculaires chez les patients présentant des événements indésirables associés au vaccin contre la COVID-19.

Tableau 2. Résultats de l’enrichissement des ensembles de gènes classés par perturbations des voies moléculaires chez les patients atteints d’un cancer nouvellement apparu après une vaccination à base d’ARN contre la COVID-19.
Afin d’explorer plus en détail le paysage moléculaire associé aux perturbations transcriptomiques induites par les vaccins, une analyse du réseau d’interactions protéine-protéine (IPP) a été réalisée pour les gènes les plus significativement dysrégulés dans chaque groupe d’étude. Comme le montre la figure 2 , la carte des IPP du groupe 1 (personnes présentant de nouveaux effets indésirables après la vaccination) et la figure 3 , celle du groupe 2 (personnes présentant un cancer après la vaccination).
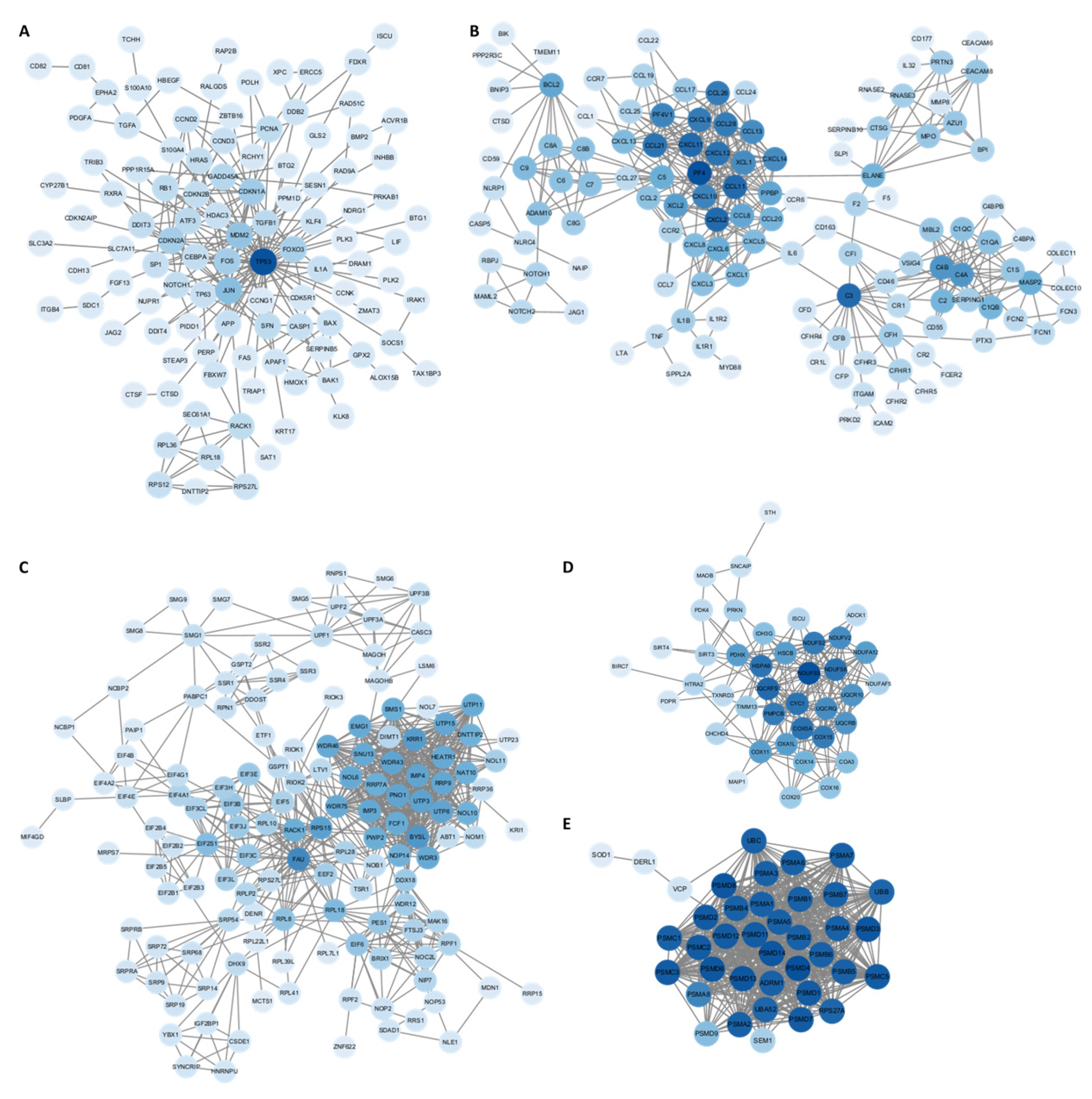
Figure 2. Réseau d’interaction protéine-protéine (IPP) des gènes les plus dérégulés du groupe 1. L’intensité de la couleur des nœuds reflète le degré d’interaction (connectivité), les nœuds plus foncés indiquant une connectivité plus élevée ou un statut de hub au sein du réseau. ( A ) Gènes différentiellement exprimés qui sont liés à la signalisation proliférative et au contrôle tumoral. ( B ) Gènes différentiellement exprimés qui sont liés à l’immunité inflammatoire systémique. ( C ) Gènes différentiellement exprimés liés à l’instabilité transcriptomique. ( D ) Gènes différentiellement exprimés qui sont liés à la mitochondrie. ( E ) Gènes différentiellement exprimés qui sont liés à la dégradation du protéasome.
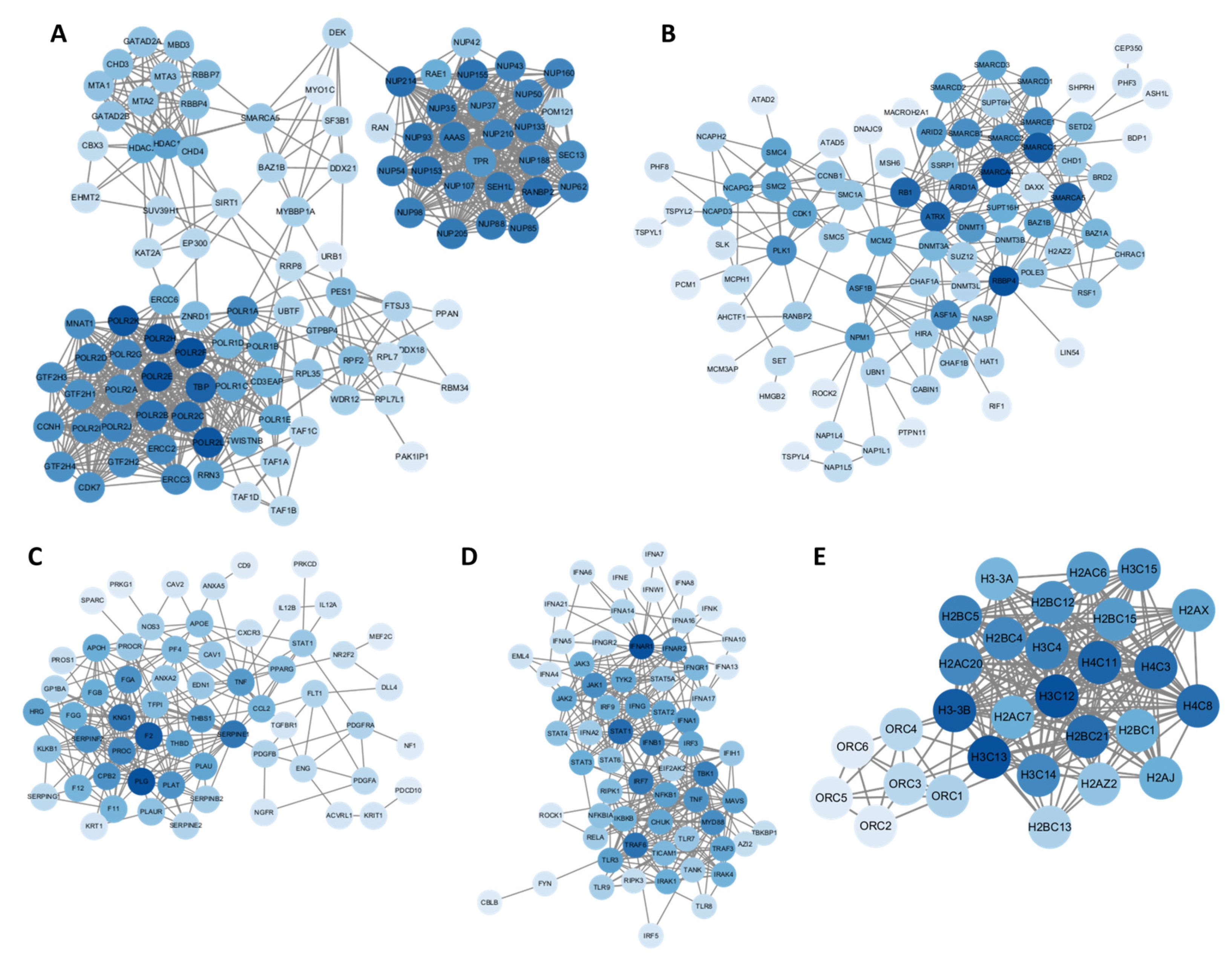
Figure 3. Réseau d’interaction protéine-protéine (IPP) des gènes les plus dérégulés du groupe 2. L’intensité de la couleur des nœuds reflète le degré d’interaction (connectivité), les nœuds plus foncés indiquant une connectivité plus élevée ou un statut de hub au sein du réseau. (A) Réseau d’interaction protéine-protéine pour les gènes régulés à la hausse qui sont liés à l’instabilité transcriptomique, au stress translationnel. (B) Réseau d’interaction protéine-protéine pour les gènes régulés à la hausse qui sont liés à l’instabilité génomique et au décalage épigénétique. (C) Réseau d’interaction protéine-protéine pour les gènes régulés à la baisse associés au dysfonctionnement endothélial. (D) Réseau d’interaction protéine-protéine pour les gènes régulés à la baisse qui sont liés à la réponse inflammatoire et immunitaire systémique. (E) Réseau d’interaction protéine-protéine pour les gènes régulés à la hausse qui sont liés à la signalisation proliférative et au contrôle tumoral supprimé.
4. Discussion
Français Le déploiement mondial rapide des vaccins à base d’ARN contre la COVID-19 a introduit une nouvelle plateforme thérapeutique impliquant des systèmes d’administration d’ARNm synthétique et de nanoparticules lipidiques (LNP). Bien qu’initialement déployés pour atténuer la propagation du SARS-CoV-2, les effets biologiques à long terme étaient inconnus. Contrairement aux vaccins conventionnels, ces formulations induisent les cellules hôtes à exprimer une glycoprotéine de pointe virale à partir de modèles d’ARNm exogènes, ce qui soulève des inquiétudes quant aux réponses cellulaires involontaires [ 32 ] . De nouveaux rapports cliniques faisant état de symptômes persistants après la vaccination, notamment de troubles neurologiques, cardiovasculaires et immunologiques, ont incité la science à examiner de près les mécanismes moléculaires potentiellement déclenchés par une exposition prolongée à l’ARNm synthétique, à ses produits de traduction et à l’activation immunitaire associée [ 2 , 3 , 33 , 34 ] . Pour étudier ces préoccupations, nous avons effectué des analyses d’enrichissement de l’ensemble des gènes (GSEA) sur les données transcriptomiques de deux cohortes de patients distinctes : les personnes présentant de nouveaux effets indésirables post-vaccinaux (groupe 1) et les patients ayant développé un cancer après la vaccination par ARNm (groupe 2). Un résumé des altérations transcriptomiques observées dans chaque groupe est illustré dans Figure 4 .
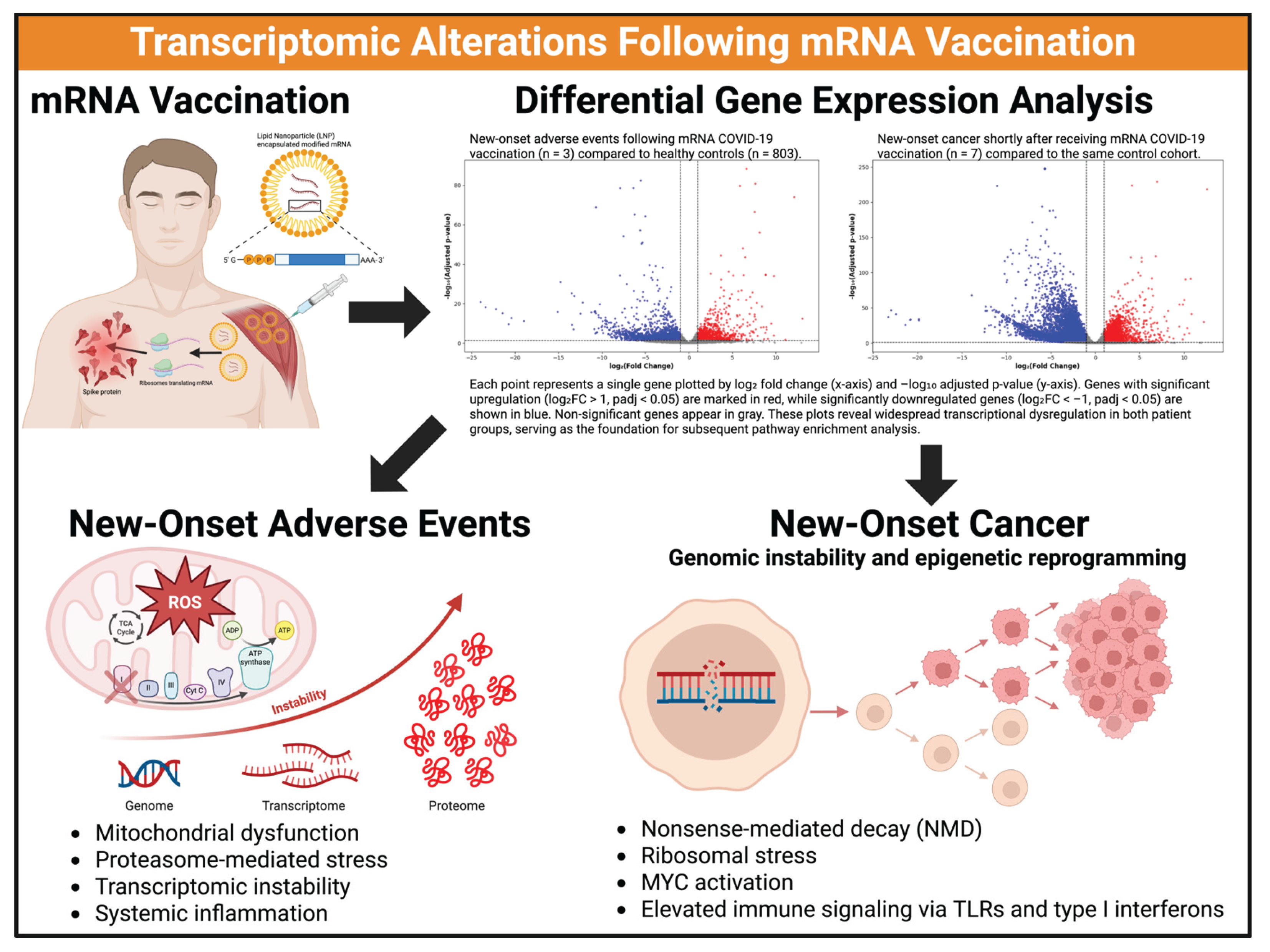
Figure 4. Altérations transcriptomiques après vaccination par ARNm. Cette illustration centrale résume le protocole expérimental, les principaux résultats et les mécanismes moléculaires proposés sous-tendant la dysrégulation transcriptomique après vaccination par ARNm synthétique contre la COVID-19. En haut à gauche : Schéma de la vaccination par ARNm, montrant l’ARNm chimiquement modifié codant pour la protéine de spicule encapsulée dans des nanoparticules lipidiques (LNP), délivré dans les cellules hôtes, initiant une traduction persistante et un engagement immunologique. En haut à droite : Les graphiques en volcan illustrent l’expression différentielle globale des gènes dans des échantillons de sang périphérique de deux cohortes affectées par rapport à des témoins sains (n = 803). À gauche : personnes présentant des effets indésirables non malins d’apparition récente (n = 3). À droite : personnes atteintes d’un cancer d’apparition récente (n = 7). Les gènes régulés à la hausse (log₂FC > 1, padj < 0,05) sont indiqués en rouge ; les gènes régulés à la baisse (log₂FC < –1, padj < 0,05) en bleu ; les gènes non significatifs en gris. En bas à gauche (événements indésirables d’apparition récente) : l’analyse transcriptomique révèle un enrichissement des voies liées au dysfonctionnement du transport des électrons mitochondriaux et aux espèces réactives de l’oxygène (ROS), au stress de dégradation des protéines médié par le protéasome, à l’activation de la surveillance de l’ARNm et à la signalisation inflammatoire systémique. En bas à droite (Cancer d’apparition récente) : Les patients atteints de cancer présentent des signes d’oncogenèse, notamment une instabilité génomique, une reprogrammation épigénétique, une dégradation médiée par le non-sens (NMD), un stress ribosomique, une signalisation proliférative induite par MYC et une activation immunitaire persistante via les récepteurs de type Toll (TLR) et les interférons de type I. Abréviations : ARNm, acide ribonucléique messager ; ROS, espèces réactives de l’oxygène ; TCA, acide tricarboxylique ; NMD, dégradation médiée par le non-sens ; TLR, récepteurs de type Toll ; MYC, oncogène de la myélocytomatose. *Créé avec BioRender.com
Français Dans la cohorte du groupe 1, l’enrichissement des ensembles de gènes liés au transport d’électrons mitochondriaux indique une perturbation transcriptionnelle dans les composants essentiels de la phosphorylation oxydative, en particulier au niveau du complexe I. La présence d’enrichissements liés à des variants impliquant PINK1, ABETA et SNCA suggère une convergence associée à une mitophagie altérée, une neurodégénérescence et un flux d’électrons défectueux. Ces résultats suggèrent que l’inflammation médiée par les pics perturbe l’homéostasie mitochondriale. En effet, des études antérieures indiquent que la protéine de pointe altère la dynamique de fusion-fission mitochondriale, supprimant la biogenèse de ses régulateurs (NRF1/2, PGC1α, TFAM) et augmentant les dommages mitochondriaux [ 35 ] . De plus, de nombreuses études ont indépendamment rapporté un dysfonctionnement mitochondrial persistant comme une caractéristique de la COVID longue [ 36 , 37 , 38 ] , soutenant davantage l’existence d’un lien mécaniste entre l’exposition à la protéine de pointe et le déséquilibre bioénergétique.
Dans la cohorte du groupe 1, l’enrichissement des ensembles de gènes associés au protéasome a indiqué l’activation de composants du système ubiquitine-protéasome. Des protéines régulées à la hausse, telles que l’ubiquitine-40S, protéine ribosomique S27a (RPS27A), la sous-unité 26S du protéasome SEM1, la polyubiquitine-B (UBB) et l’ubiquitine-60S, protéine ribosomique L40 (UBA52), ainsi que plusieurs sous-unités centrales des complexes protéasomes 20S et 19S, dont PSMA2-5, PSMA8, PSMC1 et PSMD7, reflètent une demande transcriptionnelle accrue pour la machinerie de dégradation des protéines, probablement en compensation des protéines mal repliées ou agrégées. L’enrichissement des ensembles de gènes associés aux variants liés au dysfonctionnement de VCP et de SOD1 soutient également la convergence avec la perturbation de la protéostasie, un phénomène fréquemment observé dans les maladies neurodégénératives et associées à une protéinopathie. L’un des facteurs potentiels de cette activation protéasomale soutenue est la présence prolongée d’ARNm synthétique en circulation, qui persiste au-delà de la traduction initiale et peut stimuler en permanence la production de protéines de pointe ou de produits de traduction aberrants. [ 18 , 19 , 20 ] . De plus, de nouvelles preuves suggèrent que la protéine de pointe du SARS-CoV-2 contient des domaines de type prion qui sont sujets au mauvais repliement et à l’agrégation. Ces propriétés peuvent être amplifiées dans l’expression induite par le vaccin, conduisant à un stress protéotoxique persistant et à des profils transcriptionnels de type neurodégénératif [ 10 ] .
Français L’instabilité transcriptomique et le stress translationnel étaient une caractéristique dominante dans les deux groupes de patients. L’enrichissement des ensembles de gènes liés à l’initiation de la traduction, à la biogenèse des ribosomes et à la surveillance de l’ARNm suggère une activation soutenue de la machinerie de synthèse des protéines. Dans le groupe 1, les voies régulées à la hausse telles que l’initiation de la traduction, la traduction cytoplasmique et la biogenèse des sous-unités ribosomiques indiquent un engagement persistant des ribosomes et de l’appareil traductionnel, probablement dû à l’expression prolongée de la protéine de pointe à partir d’ARNm synthétique stabilisé [ 10 , 18 , 20 ] . L’enrichissement des gènes liés à la dégradation médiée par le non-sens (NMD) chez les patients présentant des événements indésirables associés au vaccin d’apparition récente contraste avec les résultats antérieurs selon lesquels le virus SARS-CoV-2 supprime le NMD pour protéger son génome d’ARN [ 39 , 40 ] . Ceci suggère que, contrairement au génome viral, l’ARNm synthétique utilisé en vaccination pourrait plutôt provoquer une activation compensatoire des mécanismes de surveillance de l’ARN, potentiellement due à une traduction persistante ou à l’accumulation de transcrits aberrants. Les schémas opposés de régulation du NMD dans ces deux contextes suggèrent des réponses cellulaires distinctes et justifient des investigations plus approfondies. Dans le groupe 2, comme dans le premier groupe, nous avons observé un enrichissement significatif des ensembles de gènes liés à la biogenèse des ribosomes, à la traduction cytoplasmique et à la surveillance de l’ARNm. Cependant, le groupe cancéreux a présenté une activation plus prononcée des voies de stress ribosomique, en particulier celles liées à l’échappement du promoteur de l’ARN polymérase I, à la maturation de l’ARNr et à la régulation épigénétique positive de l’expression de l’ARNr, indiquant une hyperactivation des fonctions nucléolaires et un débit traductionnel élevé. Bien que ces programmes transcriptionnels soient classiquement associés aux cellules tumorales pour favoriser une prolifération incontrôlée, [ 41 , 42 , 43 ] , leur détection dans des échantillons de sang périphérique reflète probablement les conséquences systémiques d’une tumeur maligne sous-jacente, telles que des altérations immunitaires systémiques ou des réponses au stress induites par l’inflammation et la signalisation liées à la tumeur [ 44 , 45 , 46 ] . Alors que le groupe d’événements indésirables liés au vaccin a montré une activation des voies de dégradation à médiation non-sens, le groupe cancéreux a affiché des signaux supplémentaires liés à la régulation transcriptionnelle médiée par les petits ARN et à la répression épigénétique de l’activité des gènes ribosomiques. La coexistence du silençage transcriptionnel et de la production accrue d’ARNr peut refléter un stress transcriptionnel sous-jacent affectant l’équilibre cellulaire.
Français Des enrichissements liés à la caractéristique de la réponse inflammatoire et immunitaire systémique ont été identifiés dans les deux groupes analysés dans ce travail. Dans le groupe 1, les gènes régulés à la hausse les plus fortement connectés dans le réseau d’interaction protéine-protéine (PPI) comprenaient LOX, CD28, CCR7 et SELL. Des découvertes récentes démontrent que l’utilisation de N1-méthylpseudouridine dans les constructions d’ARNm peut induire un décalage du cadre ribosomique de +1, entraînant la production de protéines hors cible qui peuvent déclencher des réponses immunitaires cellulaires non intentionnelles [ 11 ] . Cette production aberrante d’antigènes pourrait représenter un nouveau mécanisme contribuant à l’inflammation systémique et à la dysrégulation immunitaire. De plus, des preuves émergentes suggèrent que la fragmentation cytoplasmique de l’ARNm dérivé du vaccin peut générer de courtes séquences d’ARN avec des propriétés de type miARN capables d’hybrider les transcrits immunitaires de l’hôte, tels que les interférons et les régulateurs anti-inflammatoires. Cette interférence post-transcriptionnelle involontaire pourrait contribuer à une dysrégulation immunitaire systémique et à des réponses inflammatoires, en particulier chez les individus présentant des comorbidités prédisposantes ou des voies de dégradation de l’ARN altérées. [ 47 ] . De plus, de nombreuses études ont déjà démontré l’activité inflammatoire des nanoparticules lipidiques d’ARNm [ 48 , 49 , 50 ] . Comparé au groupe 1, où le déséquilibre immunitaire était davantage orienté vers l’activation cellulaire aberrante et la suppression humorale, le groupe 2 présentait un schéma distinct d’activation immunitaire innée. L’analyse de l’enrichissement de l’ensemble des gènes a révélé la régulation positive des voies impliquant les interférons de type I, les récepteurs de type Toll (TLR) et la signalisation inflammatoire induite par NF–κB. L’enrichissement de l’axe RIG-I/MDA5–IRF7, TLR3/7/8/9–IRF5/7 et la cascade d’interférons JAK-STAT suggère un engagement persistant des mécanismes de détection de l’ARN et de leurs programmes transcriptionnels pro-inflammatoires en aval. Bien que ces réponses soient essentielles à l’immunité antivirale, leur activation chronique dans le sang périphérique des patients atteints de cancer pourrait être liée à l’inflammation, à l’épuisement immunitaire et à l’édition immunitaire de la tumeur [ 51 , 52 , 53 ] .
Français Pour le groupe 1, un NES négatif pour la caractéristique d’angiogenèse suggère une régulation négative transcriptionnelle ou une inhibition post-transcriptionnelle des principaux médiateurs angiogéniques. Ceci peut être lié à l’hypothèse de fragmentation de l’ARN soutenue par [ 47 ] , dans laquelle des fragments de type miRNA dérivés de l’ARNm vaccinal clivé s’hybrident avec les transcrits endothéliaux, supprimant leur traduction et contribuant au dysfonctionnement endothélial systémique et à une altération de la réparation vasculaire. Dans le groupe cancer, l’analyse transcriptomique a révélé une régulation négative significative des ensembles de gènes impliqués dans la régulation négative de la prolifération des cellules endothéliales et la régulation négative de la coagulation. La suppression de ces voies de régulation peut indiquer une activation endothéliale incontrôlée ou un décalage pro-thrombotique vasculaire, conditions qui peuvent être liées aux événements indésirables de la protéine Spike et à la progression tumorale [ 54 , 55 , 56 , 57 ] .
Pour les groupes 1 et 2, nous avons observé un enrichissement des signatures des gènes Hallmark de signalisation proliférative et de contrôle tumoral supprimé. Pour le groupe 1, un enrichissement positif des ensembles de gènes régulés par MYC suggère un programme transcriptionnel oncogène actif favorisant la croissance et l’adaptation tumorales. Ce biais prolifératif est aggravé par la régulation négative des voies suppressives de tumeurs, notamment la signature inhibée par KRAS, les réseaux de régulation p53 et les inhibiteurs de la voie Wnt. Ces résultats suggèrent un microenvironnement propice à une expansion cellulaire incontrôlée, à une perte de surveillance apoptotique et à une diminution de la réactivité aux signaux antiprolifératifs. Notamment, de nombreuses études antérieures ont rapporté que la protéine de pointe peut déclencher la voie mitogène, par la régulation négative de l’expression de l’ACE2, qui favorise une cascade de signalisation médiée par le récepteur de l’angiotensine II de type I (AT1R), induisant les molécules régulatrices transcriptionnelles facteur nucléaire-κB (NF-κB) et protéine activatrice 1 (AP-1)/c-Fos via l’activation de la protéine kinase activée par les mitogènes (MAPK) [ 58 , 59 , 60 ] . Conformément à cette voie moléculaire, nos patients étudiés du groupe 1 et du groupe 2 ont également présenté une expression d’ACE2 très sous-régulée par rapport au contrôle normal (changement log-fold : -4,3 et -4,8, respectivement). En relation avec cette caractéristique, le groupe 2 a montré un enrichissement des voies impliquées dans le processus de réplication de l’ADN, ce qui peut refléter une prolifération accrue ou un stress réplicatif dans les cellules immunitaires circulantes, potentiellement induit par des effets systémiques liés à la tumeur ou une activation immunitaire prolongée [ 51 , 52 , 53 ] .
Français Seul le groupe 2 a présenté des enrichissements liés à la caractéristique d’instabilité génomique et de décalage épigénétique. Les voies clés enrichies montrent une régulation aberrante de la modification des histones, de l’empaquetage de l’ADN et du silençage épigénétique, qui sont fréquemment observés dans les événements oncogènes. En parallèle, l’enrichissement de la voie de signalisation cGAS–STING indique une reconnaissance immunitaire innée des fragments d’ADN cytoplasmiques, un marqueur bien établi des dommages à l’ADN et de l’instabilité chromosomique [ 61 ] . Cette voie peut être liée à l’inflammation favorisant les tumeurs et à l’édition immunitaire [ 51 , 52 , 53 ] .
5. Conclusions
Cette étude fournit des preuves transcriptomiques de perturbations moléculaires chez deux populations de patients – ceux présentant des effets indésirables non malins d’apparition récente et ceux atteints de cancers nouvellement diagnostiqués – des mois à des années après la vaccination par ARNm contre la COVID-19. Grâce à l’analyse d’expression différentielle et à la GSEA, nous avons identifié des signatures caractéristiques de dysfonctionnement mitochondrial, de stress translationnel, de dérégulation immunitaire, de perturbation endothéliale et de signalisation proliférative dans les deux cohortes. Notamment, si les deux groupes partageaient des perturbations transcriptionnelles des voies immunitaires et translationnelles, le groupe cancéreux présentait des signatures supplémentaires d’instabilité génomique et de remodelage épigénétique.
L’expression persistante de la protéine Spike, l’activité prolongée de l’ARNm synthétique et les modifications de l’ARN telles que la N1-méthylpseudouridine semblent contribuer à une activité ribosomique aberrante soutenue, au stress protéostasique et à l’activation immunitaire. Nos résultats mettent également en évidence des signaux transcriptionnels indiquant des conditions favorisant la tumeur, notamment la suppression des réseaux p53, l’activation des cibles MYC et une altération de la signalisation de l’interféron, en particulier dans le contexte d’une dysrégulation épigénétique dans la cohorte cancéreuse. Ces observations suggèrent que la reprogrammation transcriptomique induite par le vaccin pourrait affecter différemment les individus, génétiquement ou immunologiquement, sur une longue période après la vaccination.
Matériel supplémentaire
Les informations complémentaires suivantes peuvent être téléchargées sur : Preprints.org.
Contributions des auteurs
NvR et WZ ont dirigé la discussion et l’interprétation des résultats. PA a préparé les ensembles de données pour l’analyse. NHKM et PM ont fourni le soutien scientifique et la supervision du projet. JC a été le chercheur principal et a supervisé la recherche. Tous les auteurs ont relu et approuvé le manuscrit final.
Financement
Cette recherche n’a reçu aucun financement externe.
Déclaration du comité d’examen institutionnel
L’étude a été menée conformément à la Déclaration d’Helsinki et approuvée par les comités d’éthique institutionnels (ou comités d’éthique) des cliniques suivantes : Rejenesis Plus Dr James Joseph IRB00014446. Forum Health Center NP Susan Harris IRB00013617. Medical Health Institute Dr Rudolph Eberwein IRB00014296. Dr Petra MD Dr Petra Davelaar IRB00014502. Humanizing Medicine Dr Mark Hancock IRB00014854.
Déclaration de consentement éclairé
Le consentement éclairé a été obtenu de tous les sujets impliqués dans l’étude
Déclaration de disponibilité des données
Nous encourageons tous les auteurs d’articles publiés dans les revues MDPI à partager leurs données de recherche. Dans cette section, veuillez indiquer où trouver les données étayant les résultats rapportés, y compris les liens vers les ensembles de données archivés publiquement, analysés ou générés pendant l’étude. Si aucune nouvelle donnée n’a été créée, ou si les données ne sont pas disponibles pour des raisons de confidentialité ou d’éthique, une déclaration est requise. Des suggestions de déclarations de disponibilité des données sont disponibles dans la section « Politiques relatives aux données de recherche MDPI » à l’ adresse https://www.mdpi.com/ethics .
Remerciements
Nous remercions le Centre de génomique de l’Université du Nord du Texas pour son soutien au séquençage, Neo7Bioscience pour son aide à l’interprétation des données et la Fondation McCullough pour sa collaboration scientifique. Nous remercions également les participants qui ont rendu cette étude possible.
Conflits d’intérêts
Cette recherche n’a bénéficié d’aucun financement public. Neo7Bioscience est une société privée de biotechnologie spécialisée dans le développement de peptides thérapeutiques personnalisés. La Fondation McCullough a apporté un soutien indépendant à la recherche scientifique. L’Université du Nord du Texas a participé au traitement des échantillons et à l’analyse transcriptomique. Les auteurs ne déclarent aucune affiliation ni implication susceptible d’influencer l’intégrité de ce travail.
Abréviations
Les abréviations suivantes sont utilisées dans ce manuscrit :
| ARNm | Acide ribonucléique messager |
| UTR | Région non traduite |
| NMD | Décomposition médiée par le non-sens |
| SV40 | Virus simien 40 |
| ORC | Complexe de reconnaissance des origines |
| ARNr | ARN ribosomique |
| cGAS | GMP-AMP synthase cyclique |
| PIQUER | Stimulateur des gènes d’interféron |
| IFN | Interféron |
| TLR | Récepteur de type Toll |
| ISG | Gène stimulé par l’interféron |
| IPP | Interaction protéine-protéine |
| DESeq2 | Séquençage d’expression différentielle (v2) |
| GSEA | Analyse d’enrichissement des ensembles de gènes |
| PBMC | Cellule mononucléaire du sang périphérique |
| PCR | Réaction en chaîne par polymérase |
| LNP | Nanoparticule lipidique |
| LIGNE 1 | Élément nucléaire 1 à longue dispersion |
Références
- Acevedo-Whitehouse, K. ; Bruno, R. Risques potentiels pour la santé liés à la vaccination à base d’ARNm : une hypothèse. Med Hypotheses 2023 , 171 , 111015. [ Google Scholar ] [ CrossRef ]
- Li, YE ; Wang, S. ; Reiter, RJ ; Ren, J. Urgences cardiovasculaires cliniques et bases cellulaires de la vaccination contre la COVID-19 : du rêve à la réalité ? Int. J. Infect. Dis. 2022 , 124 , 1–10. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Mohseni Afshar, Z. ; Tavakoli Pirzaman, A. ; Liang, JJ ; Sharma, A. ; Pirzadeh, M. ; Babazadeh, A. ; Hashemi, E. ; Deravi, N. ; Abdi, S. ; Allahgholipour, A. ; et al. Passons-nous à côté d’effets indésirables rares induits par la vaccination contre la COVID-19 ? Frontiers in Medicine 2022 , 9. [ Google Scholar ] [ CrossRef ]
- Solomon, AL ; Ratchford, EV ; Armitage, KB ; Kovacic, JC. Page d’information destinée aux patients atteints de maladies vasculaires : Considérations vasculaires liées aux vaccins contre la COVID-19. Vasc. Med. 2021 , 27 , 102–106. [ Google Scholar ] [ CrossRef ]
- Yoon, D. ; Jeon, H.-L. ; Noh, Y. ; Choe, YJ ; Choe, S.-A. ; Jung, J. ; Shin, J.-Y. Enquête nationale sur l’expérience des personnes vaccinées contre la COVID-19 à ARNm concernant les effets indésirables et les facteurs associés. J. Korean Med Sci. 2023 , 38 , e170. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Ferreira-Da-Silva, \. ; Lobo, MF ; Pereira, AM ; Morato, M. ; Polónia, JJ ; Ribeiro-Vaz, I. Analyse en réseau des profils d’événements indésirables après vaccination par des vaccins à ARNm contre la COVID-19 : données réelles issues de la base de données européenne de pharmacovigilance EudraVigilance. Front. Med. 2025 , 12 , 1501921. [ Google Scholar ] [ CrossRef ]
- Yasmin, F.; Najeeb, H.; Naeem, U.; Moeed, A.; Atif, AR; Asghar, MA-O.; Nimri, N.; Saleem, M.; Bandyopadhyay, D.; Krittanawong, C.; et al. Événements indésirables suite aux vaccins à ARNm contre la COVID-19 : une revue systématique des complications cardiovasculaires, de la thrombose et de la thrombocytopénie.
- Nyström, S. ; Hammarström, P. Amyloidogenèse de la protéine Spike du SRAS-CoV-2. Confiture. Chimique. Soc. 2022 , 144 , 8945-8950. [ Google Scholar ] [ CrossRef ]
- Manniche, V.F. ; Schmeling, M. ; Gilthorpe, J.D. ; Hansen, PR. Taux de conceptions réussies selon le statut vaccinal contre la COVID-19 : données de la République tchèque. Revue internationale des risques et de la sécurité en médecine 2025. [ Google Scholar ] [ CrossRef ]
- Seneff, S. ; Kyriakopoulos, AM ; Nigh, G. ; A. McCullough, P. ; McCullough, PA. Rôle potentiel de la protéine Spike dans les maladies neurodégénératives : revue narrative. Cureus 2023 , 15 , e34872. [ Google Scholar ] [ CrossRef ]
- Stiving, AQ ; Roose, BW ; Tubbs, C. ; Haverick, M. ; Gruber, A. ; Rustandi, RR ; Kuiper, J. ; Schombs, M. ; Schuessler, H. ; Li, X. Caractérisation de la fonctionnalité et de la fidélité de la traduction des vaccins à ARNm par détection par spectrométrie de masse basée sur une plateforme. npj Vaccines 2025 , 10 , 1–10. [ Google Scholar ] [ CrossRef ]
- Lim, S. ; Yocum, RR ; Silver, PA ; Way, JC. Taux élevés d’intégration spontanée d’ADN linéaires à extrémités modifiées lors de la transfection de cellules de mammifères. Sci. Rep. 2023 , 13 , 1–8. [ Google Scholar ] [ CrossRef ]
- Šenigl, F. ; Soikkeli, AI ; Prost, S. ; Schatz, DG ; Slavková, M. ; Hejnar, J. ; Alinikula, J. L’amplificateur du virus SV40 fonctionne comme un élément ciblant l’hypermutation somatique avec une activité tumorigène potentielle. Tumour Virus Res. 2024 , 18 , 200293. [ Google Scholar ] [ CrossRef ]
- Dean, DA; Dean Bs Fau – Muller, S.; Muller S Fau – Smith, LC; Smith, LC Exigences de séquence pour l’importation nucléaire de plasmides.
- Kämmerer, U.; Schulz, V.; Steger, K. Les injections de COVID-19 à base d’ARN de BioNTech contiennent de grandes quantités d’ADN résiduel, y compris une séquence promotrice/amplificateur SV40. 2024.
- Drayman, N. ; Ben-Nun-Shaul, O. ; Butin-Israeli, V. ; Srivastava, R. ; Rubinstein, AM ; Mock, CS ; Elyada, E. ; Ben-Neriah, Y. ; Lahav, G. ; Oppenheim, A. L’élévation du taux de p53 dans les cellules humaines interrompt l’infection par le virus SV40 en inhibant l’expression de T-ag. Oncotarget 2016 , 7 , 52643–52660. [ Google Scholar ] [ CrossRef ]
- Strayer, D. ; Branco F Fau – Zern, MA; Zern Ma Fau – Yam, P. ; Yam P Fau – Calarota, SA; Calarota Sa Fau – Nichols, CN ; Nichols Cn Fau – Zaia, JA; Zaia Ja Fau – Rossi, J.; Rossi J Fau – Li, H.; Li H Fau – Parashar, B. ; Parashar B Fau – Ghosh, S. ; et coll. Durabilité de l’expression du transgène et de l’intégration des vecteurs : vecteurs de thérapie génique recombinants dérivés du SV40.
- Patterson, BK ; Yogendra, R. ; Francisco, EB ; Guevara-Coto, J. ; Long, E. ; Pise, A. ; Osgood, E. ; Bream, J. ; Kreimer, M. ; Jeffers, D. ; et al. Détection de la protéine S1 dans les monocytes CD16+ jusqu’à 245 jours chez les personnes atteintes du syndrome post-vaccinal COVID-19 (PCVS) SARS-CoV-2-négatif. Hum. Vaccines Immunother. 2025 , 21 , 2494934. [ Google Scholar ] [ CrossRef ]
- Boros, LG ; Kyriakopoulos, AM ; Brogna, C. ; Piscopo, M. ; McCullough, PA ; Seneff, S. ARNm biochimiquement modifié de longue durée et ses protéines de pointe recombinantes décalées dans les tissus et la circulation sanguine humains après vaccination contre la COVID-19. Pharmacol. Res. Perspect. 2024 , 12 , e1218. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Yonker, LM ; Swank, Z. ; Bartsch, YC ; Burns, MD ; Kane, A. ; Boribong, BP ; Davis, JP ; Loiselle, M. ; Novak, T. ; Senussi, Y. ; et al. Protéine de spicule circulante détectée dans la myocardite post-vaccinale à ARNm COVID-19. Circulation 2023 , 147 , 867–876. [ Google Scholar ] [ CrossRef ]
- Bruttel, V.; Washburne, A.; VanDongen, A. L’empreinte endonucléase indique une origine synthétique du SARS-CoV-2. bioRxiv 2023 . [ Google Scholar ] [ CrossRef ]
- Dobin, A.; Davis Ca Fau – Schlesinger, F.; Schlesinger F Fau – Drenkow, J.; Drenkow J Fau – Zaleski, J.; Zaleski C Fau – Jha, S.; Jha S Fau – Batut, P.; Batut P Fau – Chaisson, M.; Chaisson M Fau – Gingeras, TR; Gingeras, TR STAR : aligneur RNA-seq universel ultra-rapide.
- Patro, R. ; Duggal, G. ; Love, MI ; Irizarry, RA ; Kingsford, C. Salmon fournit une quantification rapide et sensible aux biais de l’expression des transcrits. Nat. Methods 2017 , 14 , 417–419. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Love, MI ; Huber, W. ; Anders, S. Estimation modérée du repliement et de la dispersion des données RNA-seq avec DESeq2. Genome Biol. 2014 , 15 , 550. [ Google Scholar ] [ CrossRef ]
- Anders, S. ; Huber, W. Analyse d’expression différentielle pour les données de comptage de séquences. Genome Biol. 2010 , 11 , R106. [ Google Scholar ] [ CrossRef ]
- Subramanian, A. ; Tamayo, P. ; Mootha, VK ; Mukherjee, S. ; Ebert, BL ; Gillette, MA ; Paulovich, A. ; Pomeroy, SL ; Golub, TR ; Lander, ES ; et al. Analyse de l’enrichissement des ensembles de gènes : une approche fondée sur les connaissances pour l’interprétation des profils d’expression pangénomiques. Proc. Natl. Acad. Sci. États-Unis 2005 , 102 , 15545–15550. [ Google Scholar ] [ CrossRef ]
- Liberzon, A. ; Birger, C. ; Thorvaldsdóttir, H. ; Ghandi, M. ; Mesirov, JP ; Tamayo, P. Base de données des signatures moléculaires, collection de gènes Hallmark. Cell Syst. 2015 , 1 , 417–425. [ Google Scholar ] [ CrossRef ]
- Szklarczyk, D. ; Franceschini, A. ; Wyder, S. ; Forslund, K. ; Heller, D. ; Huerta-Cepas, J. ; Simonovic, M. ; Roth, A. ; Santos, A. ; Tsafou, KP ; et al. STRING v10 : Réseaux d’interactions protéine-protéine, intégrés à l’arbre du vivant. Nucleic Acids Res. 2015 , 43 , D447–D452. [ Google Scholar ] [ CrossRef ]
- Rao, V.S. ; Srinivas, K. ; Sujini, GN ; Kumar, GNS. Détection des interactions protéine-protéine : méthodes et analyse. Int. J. Proteom. 2014 , 1–12 . [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Bebek, G. Identification des réseaux d’interactions génétiques. Dans Statistical Human Genetics: Methods and Protocols, Elston, RC, Satagopan, JM, Sun, S. (dir.) ; Humana Press : Totowa, NJ, 2012 ; p. 483–494. [ Google Scholar ]
- Szklarczyk, D. ; Kirsch, R. ; Koutrouli, M. ; Nastou, K. ; Mehryary, F. ; Hachilif, R. ; Gable, AL ; Fang, T. ; Doncheva, NT ; Pyysalo, S. ; et al. La base de données STRING en 2023 : réseaux d’association protéine-protéine et analyses d’enrichissement fonctionnel pour tout génome séquencé d’intérêt. Nucleic Acids Res. 2022 , 51 , D638–D646. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Speicher, DJ, Jessica Rose, LM Gutschi, David M. Wiseman, PhD, et Kevin McKernan. Fragments d’ADN détectés dans les vaccins monovalents et bivalents Pfizer/Biontech et Moderna Modrna contre la COVID-19 en Ontario, Canada : relation dose-réponse exploratoire avec les effets indésirables graves. Prépublications OSF 2023.
- Trougakos, IP ; Terpos, E. ; Alexopoulos, H. ; Politou, M. ; Paraskevis, D. ; Scorilas, A. ; Kastritis, E. ; Andreakos, E. ; Dimopoulos, MA Effets indésirables des vaccins à ARNm contre la COVID-19 : l’hypothèse du pic. Tendances Mol. Méd. 2022 , 28 , 542-554. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Fang, L. ; Tang, T. ; Hu, M. ; Hesham, AE-L. Identification des gènes différentiellement exprimés dans la COVID-19 et analyse bioinformatique intégrée des voies de signalisation. Genet. Res. 2021 , 2021 , 1–10. [ Google Scholar ] [ CrossRef ]
- Chang, X. ; Ismail, NI ; Rahman, A. ; Xu, D. ; Chan, RWY ; Ong, S.-B. La COVID-19 longue durée et le cœur : les mitochondries cardiaques sont-elles le chaînon manquant ? Signal redox des antioxydants. 2022 , 38 , 599–618. [ Google Scholar ] [ CrossRef ]
- Molnar, T. ; Lehoczki, A. ; Fekete, M. ; Varnaï, R. ; Zavori, L. ; Erdo-Bonyar, S. ; Simon, D. ; Berki, T. ; Csecsei, P. ; Ezer, E. Dysfonctionnement mitochondrial dans les cas de COVID long : mécanismes, conséquences et approches thérapeutiques potentielles. GéroScience 2024 , 46 , 5267-5286. [ Google Scholar ] [ CrossRef ]
- Rurek, M. Mitochondries dans la COVID-19 : perspectives cellulaires et moléculaires. Front. Physiol. 2024 , 15 , 1406635. [ Google Scholar ] [ CrossRef ]
- Cao, X.; Nguyen, V.; Tsai, J.; Gao, C.; Tian, Y.; Zhang, Y.; Carver, W.; Kiaris, H.; Cui, T.; Tan, W. La protéine Spike du SARS-CoV-2 induit des perturbations transcriptionnelles à long terme des gènes métaboliques mitochondriaux, provoque une fibrose cardiaque et réduit la contractilité myocardique chez les souris obèses. Mol. Metab. 2023 , 74 , 101756. [ Google Scholar ] [ CrossRef ]
- Mallick, M. ; Boehm, V. ; Xue, G. ; Blackstone, M. ; Gehring, NH ; Chakrabarti, S. Modulation de l’activité catalytique d’UPF1 lors de l’interaction de la protéine de la nucléocapside du SARS-CoV-2 avec des facteurs impliqués dans la dégradation de l’ARNm médiée par des ARNm non-sens. Nucleic Acids Res. 2024 , 52 , 13325–13339. [ Google Scholar ] [ CrossRef ]
- Nuccetelli, V. ; Mghezzi-Habellah, M. ; Deymier, S. ; Roisin, A. ; Gérard-Baraggia, F. ; Rocchi, C. ; Coureux, P.-D. ; Gouet, P. ; Cimarelli, A. ; Mocquet, V. ; et al. La protéine de la nucléocapside du SARS-CoV-2 interfère avec l’activation enzymatique complète d’UPF1 et son interaction avec UPF2. Nucleic Acids Res. 2025 , 53 . [ Google Scholar ] [ CrossRef ]
- Pitts, S. ; Laiho, M. Régulation de la stabilité et de la fonction de l’ARN polymérase I. Cancers 2022 , 14 , 5776. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Ferreira, R. ; Schneekloth, JS ; Panov, KI ; Hannan, KM ; Hannan, RD. Le ciblage de la transcription de l’ARN polymérase I pour le traitement du cancer atteint sa maturité. Cells 2020 , 9 , 266. [ Google Scholar ] [ CrossRef ]
- López, J. ; Blanco, S. Exploration du rôle des modifications de l’ARN ribosomique dans le cancer. Curr. Opin. Genet. Dev. 2024 , 86 , 102204. [ Google Scholar ] [ CrossRef ]
- Abusanad, A. Utilisation d’un biomarqueur inflammatoire du sang périphérique (PBIB) pour prédire la réponse au traitement systémique chez les patientes atteintes d’un cancer du sein. J. Fam. Med. Prim. Care 2023 , 12 , 3368–3373. [ Google Scholar ] [ CrossRef ]
- Huai, Q. ; Luo, C. ; Song, P. ; Bie, F. ; Bai, G. ; Li, Y. ; Liu, Y. ; Chen, X. ; Zhou, B. ; Sun, X. ; et al. La dynamique des biomarqueurs inflammatoires du sang périphérique reflète la réponse au traitement et prédit le pronostic chez les patients atteints d’un cancer du poumon non à petites cellules sous immunothérapie néoadjuvante. Cancer Sci. 2023 , 114 , 4484–4498. [ Google Scholar ] [ CrossRef ]
- Xie, Y. ; Yu, Q. ; Zhu, Y. ; Wu, W. ; Xiao, R. ; Wang, N. ; Zhu, L. ; Li, P. ; Chen, T. Intérêt des marqueurs d’inflammation du sang périphérique dans l’évaluation du risque et la prédiction du cancer du poumon. Futur. Sci. OA 2025 , 11 , 2476870. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Demongeot, J. ; Fougère, C. Vaccins à ARNm contre la COVID-19 : faits et hypothèses sur la fragmentation et l’encapsulation. Vaccines 2022 , 11 , 40. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Kiaie, SH ; Zolbanin, NM ; Ahmadi, A. ; Bagherifar, R. ; Valizadeh, H. ; Kashanchi, F. ; Jafari, R. Progrès récents dans les thérapies à base d’ARNm-LNP : aspects immunologiques et pharmacologiques. J. Nanobiotechnology 2022 , 20 , 1–20. [ Google Scholar ] [ CrossRef ]
- Parry, PI ; Lefringhausen, A. ; Turni, C. ; Neil, CJ ; Cosford, R. ; Hudson, NJ ; Gillespie, J. « Spikeopathie » : la protéine Spike de la COVID-19 est pathogène, d’après l’ARNm du virus et du vaccin. Biomedicines 2023 , 11 , 2287. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Sharma, P. ; Hoorn, D. ; Aitha, A. ; Breier, D. ; Peer, D. Nature immunostimulante des nanoparticules lipidiques d’ARNm. Adv. Drug Deliv. Rev. 2024 , 205 , 115175. [ Google Scholar ] [ CrossRef ]
- Hu, A. ; Sun, L. ; Lin, H. ; Liao, Y. ; Yang, H. ; Mao, Y. Exploiter les voies immunitaires innées pour des avancées thérapeutiques dans le cancer. Transducteur de signal. Cible. Thérapie. 2024 , 9 , 1–59. [ Google Scholar ] [ CrossRef ]
- Maiorino, L. ; Daßler-Plenker, J. ; Sun, L. ; Egeblad, M. Immunité innée et physiopathologie du cancer. Annu. Rev. Pathol. Mech. Dis. 2021 , 17 , 425–457. [ Google Scholar ] [ CrossRef ]
- Yi, M. ; Li, T. ; Niu, M. ; Mei, Q. ; Zhao, B. ; Chu, Q. ; Dai, Z. ; Wu, K. Exploitation de l’immunité innée pour l’immunothérapie du cancer. Mol. Cancer 2023 , 22 , 1–55. [ Google Scholar ] [ CrossRef ]
- Blann, AD. Marqueurs d’activation des cellules endothéliales dans le cancer. Thromb. Res. 2012 , p. 129 , S122–S126. [ Google Scholar ] [ CrossRef ]
- Smeda, M. ; Przyborowski, K. ; Stojak, M. ; Chlopicki, S. La barrière endothéliale et les métastases cancéreuses : l’aspect protecteur de la fonction plaquettaire est-il important ? Biochem. Pharmacol. 2020 , 176 , 113886. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Zheng, Y. ; Zhao, J. ; Li, J. ; Guo, Z. ; Sheng, J. ; Ye, X. ; Jin, G. ; Wang, C. ; Chai, W. ; Yan, J. ; et al. La protéine Spike du SARS-CoV-2 provoque une coagulation sanguine et une thrombose par liaison compétitive à l’héparane sulfate. Int. J. Biol. Macromol. 2021 , 193 , 1124–1129. [ Google Scholar ] [ CrossRef ]
- Becker, RC ; Tantry, États-Unis ; Khan, M. ; Gurbel, PA. Thrombus lié à la COVID-19 : distinction des caractéristiques pathologiques, mécanistiques et phénotypiques et prise en charge. J. Thromb. Thrombolyse 2024 , 58 , 15–49. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Angues, RV ; Bustos, YP. Vaccination contre le SARS-CoV-2 et hypothèse multi-hit de l’oncogenèse. Cureus 2023 , 15 , e50703. [ Google Scholar ] [ CrossRef ]
- Patra, T. ; Meyer, K. ; Geerling, L. ; Isbell, TS ; Hoft, DF ; Brien, J. ; Pinto, AK ; Ray, RB ; Ray, R. ; Lee, B. La protéine de spicule du SARS-CoV-2 favorise la signalisation trans de l’IL-6 par activation de la signalisation du récepteur de l’angiotensine II dans les cellules épithéliales. PLOS Pathog. 2020 , 16 , e1009128. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Suzuki, YJ ; Gychka, SG. La protéine Spike du SARS-CoV-2 déclenche la signalisation cellulaire dans les cellules hôtes humaines : implications pour les conséquences possibles des vaccins contre la COVID-19. Vaccines 2021 , 9 , 36. [ Google Scholar ] [ CrossRef ]
- Kwon, J. ; Bakhoum, SF. La voie cGAS–STING de détection de l’ADN cytosolique dans le cancer. Cancer Discov. 2020 , 10 , 26–39. [ Google Scholar ] [ CrossRef ] [ PubMed ]
| Avis de non-responsabilité/Note de l’éditeur : Les déclarations, opinions et données contenues dans toutes les publications sont uniquement celles des auteurs et contributeurs individuels et non celles de MDPI et/ou du ou des éditeurs. MDPI et/ou le ou les éditeurs déclinent toute responsabilité pour toute blessure aux personnes ou aux biens résultant des idées, méthodes, instructions ou produits mentionnés dans le contenu. |
Droits d’auteur : Cet article en libre accès est publié sous une licence Creative Commons CC BY 4.0 , qui autorise le téléchargement, la distribution et la réutilisation gratuits, à condition que l’auteur et la préimpression soient cités dans toute réutilisation.
